Estudios de Diagnóstico Prenatal en 2.696 Casos. Avances en la Identificación de las Causas de Malformaciones Fetales por Estudios Moleculares
Autores:
Ratto C, Hirst M, Rodríguez A, Petracchi F, Igarzabal L. (1)
Introducción
La ecografía puede detectar anomalías estructurales fetales desde el primer trimestre en adelante. Estas anomalías pueden estar indicando una patología genética de base, pero muchas veces arribar a un diagnóstico certero puede ser complejo.
Históricamente, el diagnóstico prenatal consistía en la detección de anomalías cromosómicas por cariotipo convencional. Luego, la introducción del microarray, permitio la detección de variaciones en el número de copias submicroscópicas, esto aumentó la detección de anomalías de cromosomas en un 3-5%. Las anomalías estructurales fetales se pueden asociar tanto a aneuploidías, como a CNVs (variantes del número de copias), como así también a enfermedades de genes únicos, para esto último es necesaria la secuenciación del ADN fetal, mediante un exoma.
El exoma analiza el 1- 2% del genoma, que codifica para proteínas, esto se puede llevar a cabo ya sea secuenciando el exoma completo, o guiado por fenotipo, utilizando paneles de genes. El rédito diagnóstico del exoma, por sobre el cariotipo convencional/microarray es de 31% aproximadamente, en las diferentes publicaciones, esto ha incrementado el número de patologías que pueden ser diagnosticadas en la etapa prenatal.
El objetivo de nuestro trabajo es describir los resultados de los casos de fetos con anomalías ecográficas y/o TN aumentada, y cariotipo euploide, que consultaron en la sección de Genética de CEMIC y que realizaron posteriormente estudios moleculares.
Objetivos
Describir los resultados de estudios moleculares prenatales en una serie de casos de fetos con cariotipo normal y malformaciones ecográficas y/o translucencia nucal (TN) > p95.
Materiales y métodos
Tipo de estudio: observacional retrospectivo y analitico.
Población: Se incluyeron 2.696 estudios invasivos para cariotipo fetal, realizados en la Sección de Genética Médica de CEMIC, Centro de Educación Médica e Investigaciones Clínicas “Norberto Quirno”, Buenos Aires, Argentina entre 2014 - 2024. La indicación del estudio invasivo para cariotipo fetal fue malformaciones ecográficas y/o translucencia nucal >p95.
Criterios de inclusión: Se seleccionaron casos con cariotipo euploide que realizaron otros estudios moleculares: microarray, secuenciación de panel de genes y exoma completo. Se analizaron los resultados. Los casos se buscaron en la base de datos de estudios prenatales invasivos de la sección, que se registran diariamente, y se completó la información a partir de las historias clínicas de dichos casos.
Criterios de exclusión: Se excluyeron aquellas casos que tuvieron un resultado aneuploide en el cariotipo, o que tuvieron resultado euploide y no continuaron realizando estudios.
Todos los casos fueron evaluados previamente al estudio invasivo con ecografías realizada por operadores experimentados utilizando un ecógrafo Samsung HS70. Se registraron y analizaron los datos de la ecografía fetal.
Análisis estadístico: Se expresarán con porcentaje (%) las variables categóricas. Se calcularon intervalos de confianza.
Resultados
De 1.766 casos con cariotipo normal, 156 (8.8%) realizaron estudios moleculares: 113 microarray, y 49 estudios de secuenciación: 24 paneles de genes guiados por fenotipo (9 rasopatías, 8 displasias esqueléticas, 3 ciliopatías, 1 holoprosencefalia, 1 hidrocefalia, 1 hidrops, 1 VACTERL), 19 exomas, 6 variantes familiares. Todos los casos de microarray/secuenciación presentaban malformaciones ecográficas y/o translucencia nucal (TN) >p95 al momento del diagnóstico. En 14 paneles y 9 exomas se realizó secuenciación con variación en el número de copias (CNVs). En 6 casos, simultáneamente microarray y panel (n=1) o exoma (n=5). En el grupo que realizó sólo microarray, se obtuvo un resultado anormal en el 8.8% (IC95% 4,3-15.6%) (10/113) y en 2.6% (IC95% 0.55-7,5%) (3/113) una variante de significado incierto (VUS). En el grupo de panel/exoma, se observó una variante patogénica que explica el fenotipo en 53.4% (IC 95% 37.6-78.8%) (23/43) de los casos: 70.8% (IC 95% 48.9-87.3%) (17/24) en los paneles con 4% VUS (IC95% 0,11-21.1%) (1/24), y 31.5% (IC95%: 12.5-56.5%) (6/19) en exomas con 36.8% (IC 95% 16.2-61.6%) (7/19) VUS. De estas variantes, 11 fueron consideradas “de novo” (10 dominantes y 1 recesiva) y 7 heredadas de padres portadores (4 recesivas y 2 dominante). En 2 paneles se diagnosticó CNV. 4
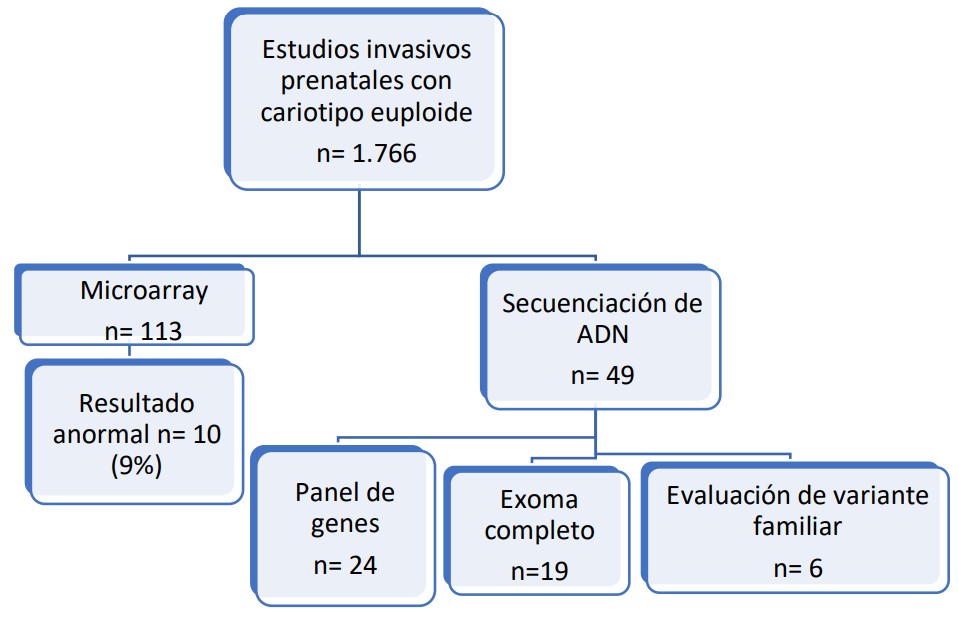
Figura 1. Estudios moleculares en muestras prenatales con cariotipo euploide
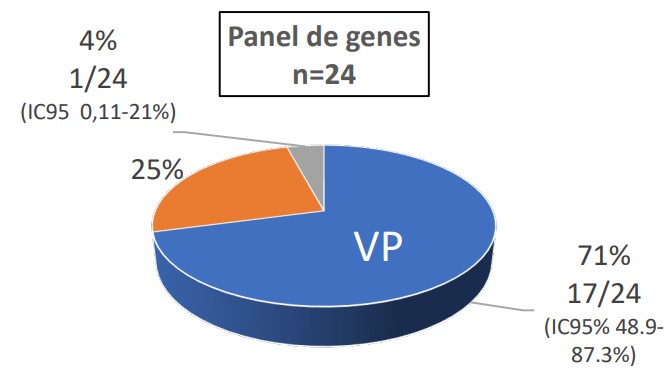
Figura 2. Resultados de paneles de genes. En color gris porcentaje (4%) (IC95% 0,11- 21%) de VUS (variantes de significado clinico incierto), color naranja resultados negativos (25%), en color azul variantes patogenicas (71%) (IC95% 48.9-87.3).

Figura 3. Resultados de exomas prenatales. En color gris VUS (variantes de significado clinico incierto) 37% (IC95%16-61%), en color naranja resultados negativos 32%, en color azul variantes patogenicas 31% (IC95%12-56%).
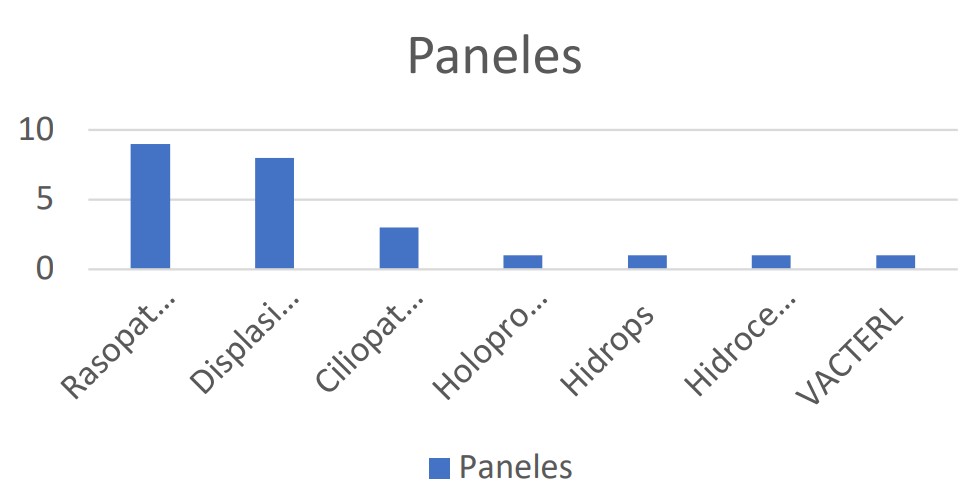
Figura 4. Tipos de paneles solicitados. 9 rasopatías, 8 displasias esqueléticas, 3 ciliopatías, 1 holoprosencefalia, 1 hidrocefalia, 1 hidrops, 1 VACTERL.
Discusion
A partir de los estudios moleculares de fetos con malformaciones o TN aumentada, con un cariotipo normal, pudo obtenerese un diagnostico etiologico en 1 cada 4 casos. En casi la mitad se interpretó el hallazgo “de novo” con bajo riesgo de recurrencia, y en un tercio con alto riesgo de recurrencia en futuros embarazos (25% o 50%). Si bien no puede determinarse el rédito diagnóstico del microarray/panel/exoma por el sesgo de selección de la muestra, los resultados de este trabajo remarcan la importancia de continuar los estudios genéticos en fetos con anomalías y cariotipo normal, para un diagnostico certero y asesoramiento preciso de pronóstico y riesgo de recurrencia.
Bibliografia
- Mellis R, Oprych K, Scotchman E, Hill M, Chitty LS. Diagnostic yield of exome sequencing for prenatal diagnosis of fetal structural anomalies: A systematic review and meta-analysis. Prenat Diagn. 2022;42: 662–685.
- Pratt M, Garritty C, Thuku M, Esmaeilisaraji L, Hamel C, Hartley T, et al. Application of exome sequencing for prenatal diagnosis: a rapid scoping review. Genet Med. 2020;22: 1925–1934.
- Lord J, McMullan DJ, Eberhardt RY, Rinck G, Hamilton SJ, Quinlan-Jones E, et al. Prenatal exome sequencing analysis in fetal structural anomalies detected by ultrasonography (PAGE): a cohort study. Lancet. 2019;393: 747–757.
- Diderich KEM, Romijn K, Joosten M, Govaerts LCP, Polak M, Bruggenwirth HT, et al. The potential diagnostic yield of whole exome sequencing in pregnancies complicated by fetal ultrasound anomalies. Acta Obstet Gynecol Scand. 2021;100: 1106–1115.
(1) Sección Genética – Departamento de Ginecología y Obstetricia –
Centro de Educación Médica e Investigaciones Clínicas – Instituto Universitario CEMIC.
Ciudad Autónoma de Buenos Aires.